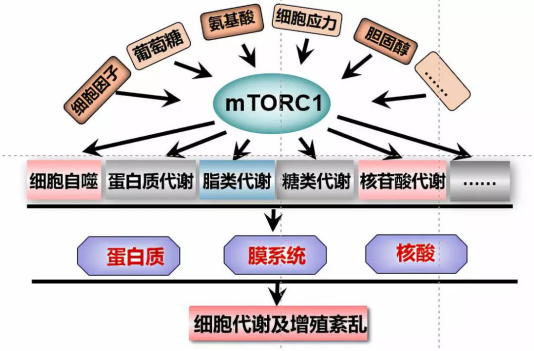
mTORC1作为微环境中营养信号的感应器,它能够感应微环境中的氨基酸、生长因子、葡萄糖、胆固醇等信号,进而调控细胞及机体内几个关键的过程:糖代谢、蛋白质代谢,脂类代谢以及细胞自噬等【1, 2】。但是,当mTORC1信号通路的关键蛋白(mTOR、GATOR、PTEN、TSC、LKB、AMPK等)发生突变,将会导致mTORC1的活性失调,进而导致细胞代谢及细胞增殖的紊乱,这也将是许多代谢相关疾病主要诱因【3, 4】。由此可见,mTORC1信号通路是维持机体代谢平衡的核心,也是细胞代谢的重要前沿领域。深入探索mTORC1信号通路的调控机制,能够为临床上基于代谢相关疾病的分子诊断、精确分型、预后分析以及靶向治疗提供重要的理论。
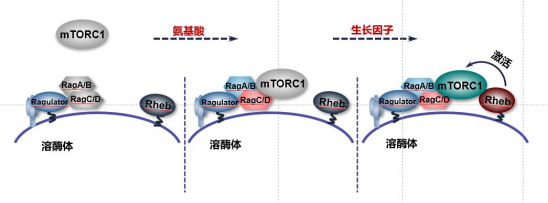
mTORC1的激活主要由氨基酸和生长因子信号协同作用,其中氨基酸信号主要通过Rags复合物调控mTORC1在溶酶体上的定位,而生长因子信号主要是通过激活Rheb进而促进mTORC1的活性【2, 5】。
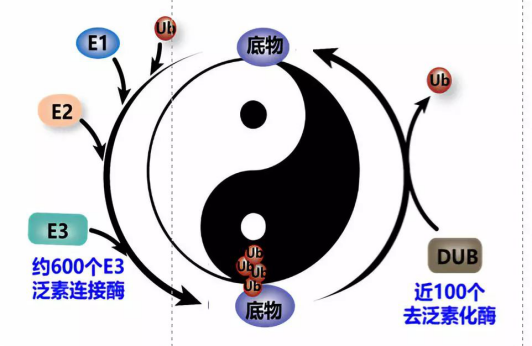
同济大学医学院王平教授课题组长期围绕肿瘤细胞与微环境交互作用机制这一肿瘤生物学关键科学问题,系统探讨了肿瘤微环境的泛素化调控。大量的证据表明泛素化修饰在肿瘤发生中有重要作用。 泛素化(包括类泛素化) 作为重要的蛋白质翻译后修饰,一直是肿瘤生物学研究的重点和热点。泛素化修饰是一个可逆的酶级联反应,由泛素连接酶和去泛素化酶等精确调控。迄今有近600种E3泛素连接酶和100种去泛素化酶被报道【6-8】。但是大部分E3泛素连接酶和去泛素化酶与肿瘤发生的关系并不清楚。
那么,泛素化是否参与了mTORC1感应微环境中的营养信号,围绕这一科学问题,同济大学医学院王平课题组以及MD Anderson Cancer Center的Hui-KuanLin课题组在2015年“背靠背”发表论文,同时发现了泛素化修饰能够通过RagA参与到mTORC1感应微环境中的氨基酸信号【9, 10】,2017年来自哈佛医学院的魏文毅教授发现泛素化修饰能够通过GβL参与到mTORC1和mTORC2信号通路的协同调控中【11】;此外,2018年,北京大学分子医学研究所/北大-清华生命科学联合中心刘颖课题组发现泛素化修饰通过降解DEPDC5参与到mTORC1感应微环境中的氨基酸信号【12】。
近日,王平课题组在Cell Research杂志上发表了题为Ubiquitination of Rheb governs growth factor-induced mTORC1 activation的研究论文,该文阐述了泛素化修饰能够通过Rheb参与到mTORC1感应微环境中的生长因子信号。

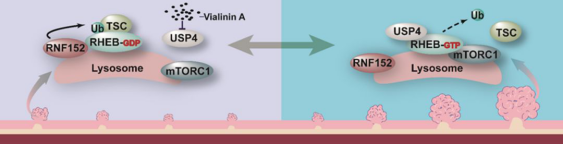
Rheb作为mTORC1的直接激活者,对于mTORC1的激活起着关键的作用【13】。该研究发现Rheb不仅可以被泛素化修饰,而且它的泛素化修饰还受生长因子信号的调控,通过筛选鉴定了它的E3泛素连接酶RNF152,去泛素化酶USP4,以及泛素化位点K8(下图)。
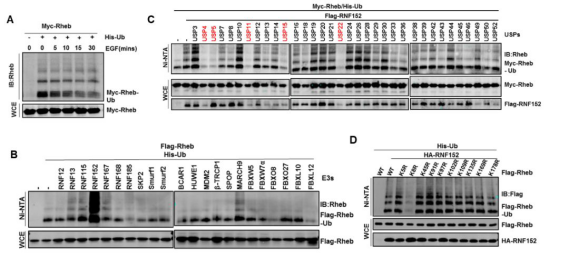
泛素化修饰作为一种翻译后修饰,它能够调控底物的稳定性、活性或者是定位,该研究发现Rheb的泛素化修饰不会影响Rheb的蛋白稳定性。通过检测MEF中RNF152和USP4的活性,发现泛素化修饰能够抑制Rheb的小G蛋白酶活性。进一步的研究发现,该泛素化修饰主要是通过促进Rheb与TSC2结合进而抑制Rheb的小G蛋白酶活性。
同时,该研究还通过RNF152、USP4的MEF细胞以及K8R knockin的细胞系,发现Rheb的泛素化修饰能够调控mTORC1的活性、细胞增殖、细胞大小以及细胞自噬等过程。此外,该研究还通过裸鼠皮下成瘤以及AOM-DSS诱导的肠癌模型,发现了Rheb的泛素化修饰在肿瘤的发生发展过程中发挥着重要的作用。
总之,该工作揭示了泛素化修饰能够通过Rheb参与到mTORC1感应微环境中的生长因子信号,阐述了Rheb的泛素化修饰是肿瘤发生发展的重要调控机制(下图)。
据悉,王平教授课题组邓露、陈蕾为本文共同第一作者,王平教授为通讯作者。
参考文献
1. Saxton, R.A. and D.M. Sabatini, mTOR Signaling in Growth, Metabolism, and Disease. Cell, 2017. 169(2): p. 361-371.
2. Dibble, C.C. and B.D. Manning, Signal integration by mTORC1 coordinates nutrient input with biosynthetic output. Nat Cell Biol, 2013. 15(6): p. 555-64.
3. Laplante, M. and D.M. Sabatini, mTOR signaling in growth control and disease. Cell, 2012. 149(2): p. 274-93.
4. Guertin, D.A. and D.M. Sabatini, Defining the role of mTOR in cancer. Cancer Cell, 2007. 12(1): p. 9-22.
5. Dibble, C.C. and L.C. Cantley, Regulation of mTORC1 by PI3K signaling. Trends Cell Biol, 2015. 25(9): p. 545-55.
6. Komander, D. and M. Rape, The ubiquitin code. Annu Rev Biochem, 2012. 81: p. 203-29.
7. Rajalingam, K. and I. Dikic, SnapShot: Expanding the Ubiquitin Code. Cell, 2016. 164(5): p. 1074-1074 e1.
8. Heride, C., S. Urbe, and M.J. Clague, Ubiquitin code assembly and disassembly. Curr Biol, 2014. 24(6): p. R215-20.
9. Deng, L., et al., The ubiquitination of rag A GTPase by RNF152 negatively regulates mTORC1 activation.Mol Cell, 2015. 58(5): p. 804-18.
10. Jin, G., et al., Skp2-Mediated RagA Ubiquitination Elicits a Negative Feedback to Prevent Amino-Acid-Dependent mTORC1 Hyperactivation by Recruiting GATOR1. Mol Cell, 2015. 58(6): p. 989-1000.
11. Wang, B., et al., TRAF2 and OTUD7B govern a ubiquitin-dependent switch that regulates mTORC2 signalling. Nature, 2017. 545(7654): p. 365-369.
12. Chen, J., et al., KLHL22 activates amino-acid-dependent mTORC1 signalling to promote tumorigenesis and ageing. Nature, 2018. 557(7706): p. 585-589.
13. Aspuria, P.J. and F. Tamanoi, The Rheb family of GTP-binding proteins. Cell Signal, 2004. 16(10): p. 1105-12.